En un artículo publicado recientemente (ver referencia al final), se describe que el SARS-CoV-2 muta mucho más lentamente que la gripe estacional. En este artículo se refiere que el SARS-CoV-2 parece tener una tasa de mutación de menos de 25 mutaciones por año, mientras que la gripe estacional tiene una tasa de mutación de casi 50 mutaciones por año.
El genoma del SARS-CoV-2 es casi dos veces más grande que el genoma de la gripe estacional, se ha estimado que la gripe estacional muta aproximadamente cuatro veces más rápido que el SARS-CoV-2. El hecho de que la gripe estacional mute tan rápido es lo que explica su capacidad para evadir nuestras vacunas. Por lo anterior la tasa de mutación significativamente más lenta del SARS-CoV-2 nos da esperanza para el desarrollo potencial de una vacuna eficaz de larga duración contra el virus.
Para entender cómo muta el virus SARS-CoV-2 debemos traer a la mente nuestras primeras lecciones de biología y partir de que este virus tiene ácido ribonucleico (ARN). El ARN es una molécula similar al ADN, y es esencialmente una copia temporal de un segmento corto de ADN. Si recordamos nuestras clases el dogma central establece que el ADN se transcribe en ARN. Actualmente la tecnología que empleamos para la secuenciación se realiza con base en el ADN. Para ello la solución es primero transcribir en reversa el ARN del virus en ADN complementario (ADNc), que luego puede secuenciarse con la tecnología actual disponible.
Pero cómo pudieron determinar lo descrito acerca de la tasa de mutación con base en su evolución. Para ello se usan modelos de evolución de su secuencia para predecir la historia del virus, y podemos usar esto para responder preguntas tales como; ¿Qué tan rápido ocurren las mutaciones? o ¿Dónde en el genoma ocurren las mutaciones? Saber qué genes están mutando con frecuencia puede ser útil en el diseño de fármacos.
Para el caso de la enfermedad COVID-19 asociada a SARS-CoV-2, existe una iniciativa global para compartir genomas virales con todos los científicos. Al existir una colección de secuencias con fechas en que se tomó la muestra así como la locación, los científicos pueden inferir el historial evolutivo de las muestras en tiempo real y utilizar la información para inferir el historial de transmisiones.
Una de esas iniciativas es Nextstrain, un proyecto de código abierto que proporciona a los usuarios informes en tiempo real sobre la propagación de la gripe estacional, el ébola y muchas otras enfermedades infecciosas. Recientemente han encabezado el seguimiento evolutivo de COVID-19 al proporcionar un análisis en tiempo real, así como un informe de situación destinado a ser legible por el público en general. Además, permite que la población mundial se beneficie de sus esfuerzos al traducir el informe de situación a muchos otros idiomas.
A continuación tomamos de la página descrita en referencias el árbol filogenético del SAR-CoV-2 que en colores nos muestra como ha mutado el virus en función del tiempo y su localización.
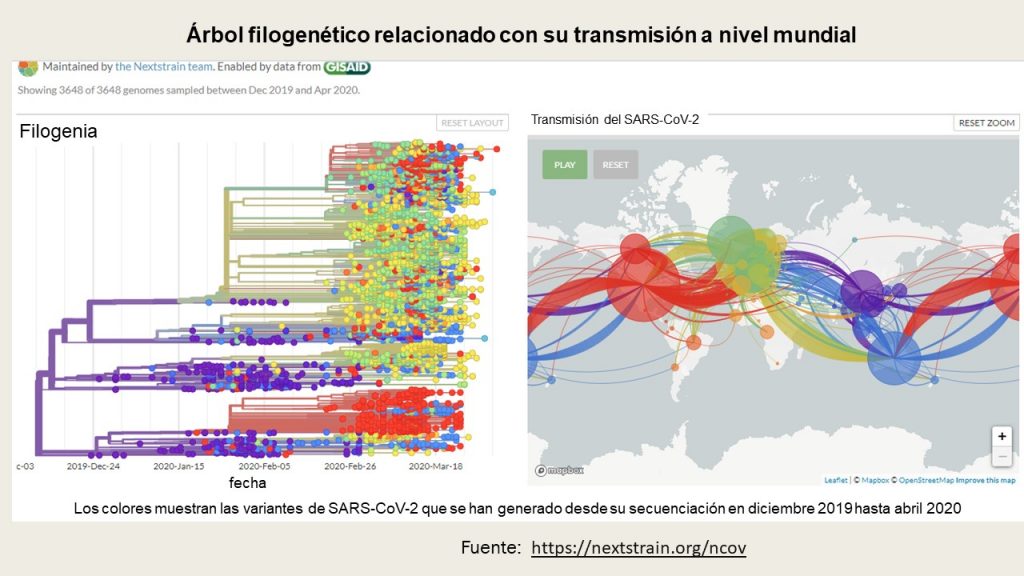
Otra imagen muy ilustrativa nos muestra como ha cambiado al ir migrando a los países.
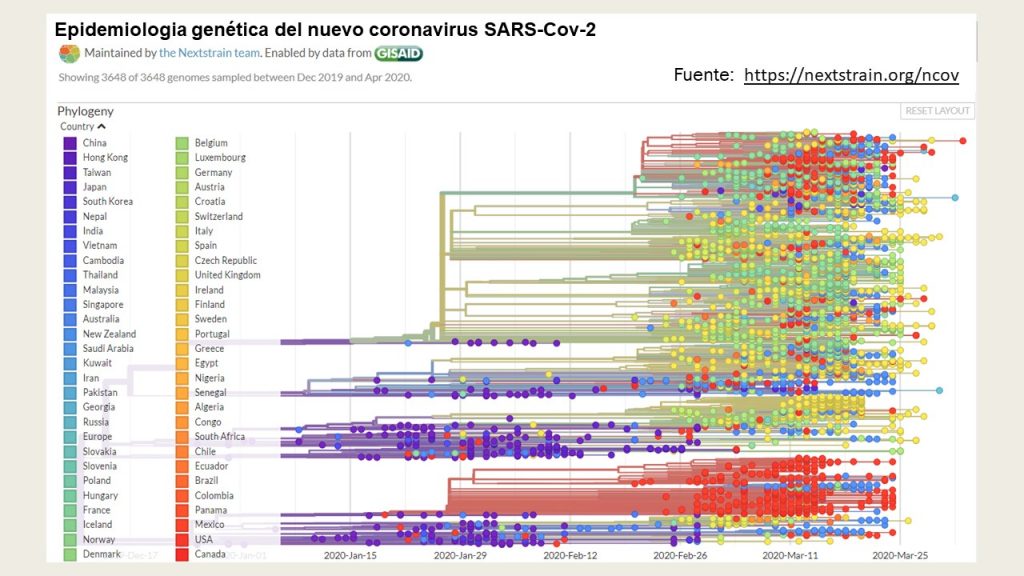
Es increíble el avance tecnológico que hemos tenido estos años para lograr en un tiempo tan corto de unos meses, que el humano ya tenga disponible esta información.
Referencias
https://www.livescience.com/coronavirus-mutation-rate.html
https://www.eldiario.es/sociedad/cambian-virus_0_1004749646.html

Felicidades Elías, decían que muto el virus, por supuesto, pero nunca imaginé que de esa manera!!
Gracias por tus artículos
Saludos
Gracias Martha Silvia lo bueno es que la tecnología y varios esfuerzos internacionales permiten visibilizar esto muy rápido. Apenas leí un artículo que compartí en redes de que realmente son tres tipos de Coronavirus los que han generado esta pandemia.